
許全智 (Hsu, Chuan-Chih )
研究助技師
- 美國普渡大學生物化學系 博士
- 蛋白質體核心實驗室
- cchsu@gate.sinica.edu.tw
- cchsu@as.edu.tw
- +886-2-2787-1157 (Office: A227, A226)
- Academia Sinica Archive
- ORCID
- Web of Science (WOS)
- Google Scholar
蛋白質轉譯後修飾是植物細胞中的重要分子事件,參與蛋白質摺疊和酵素激活的過程。其中,N-糖基化和磷酸化尤為關鍵,因為它們調節植物生長和對環境逆境的反應。然而,對這兩個修飾之間複雜交互作用以及它們對激酶活性和下游信號傳導的集體影響的全面理解仍然是一個未解之謎。質譜儀已成為系統性分析磷酸化和N-糖基化的關鍵工具,它能夠鑑定修飾位點並解開N-糖基鏈的結構。然而,修飾胜肽的低豐度和N-糖基化的異質性等挑戰妨礙了這些胜肽的直接質譜檢測。
蛋白質體學核心實驗室的研究興趣是開發新穎蛋白質體學分析策略,特別是N-糖基化和磷酸化胜肽富集方法,目的在揭示植物在環境逆境下依賴磷酸化和N-糖基化的信號傳導。我們實驗室在開發植物磷酸化和N-糖基化胜肽富集的樣本前處理工作流程方面具有專業知識。我們最近取得了一些進展:
一、基於懸浮捕捉法的植物磷酸蛋白質體學樣本前處理流程
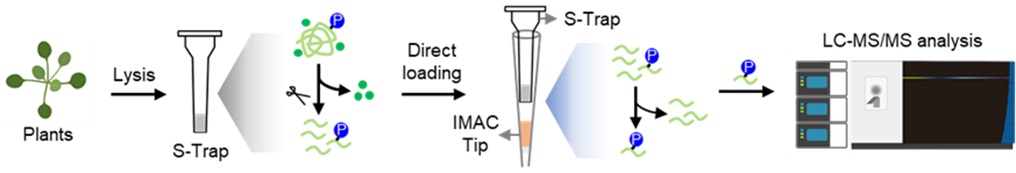
質譜磷酸化蛋白質體學已轉變了我們研究植物磷酸化驅動信號傳導的方式。然而,儘管普遍使用蛋白質沉澱法來去除污染物並提高樣品純度,這種方法限制了植物磷酸蛋白質體學的靈敏度和分析通量。在這項研究中,我們建立了一種創新方法,在樣本前處理工作流程中用懸浮捕捉法取代了蛋白質沉澱法,稱為串聯懸浮捕捉與固定金屬離子親和層析法。這種設計結合了懸浮捕捉微管柱和鐵離子固定金屬離子親和層析微量滴管。與傳統蛋白質沉澱方法進行比較,串聯懸浮捕捉與固定金屬離子親和層析法提高了超過百分之三十的阿拉伯芥磷酸化蛋白體覆蓋率。將串聯懸浮捕捉與固定金屬離子親和層析方法應用於研究阿拉伯芥的離層素磷酸化訊息信號,揭示了離層素誘導多重磷酸化胜肽,這代表了它們在早期離層素信號傳導中的重要性。
二、同時高通量植物磷酸化蛋白質體學和N-糖基化蛋白質體學的流線型串聯固定金屬離子親和層析與親水交互作用液相層析法流程
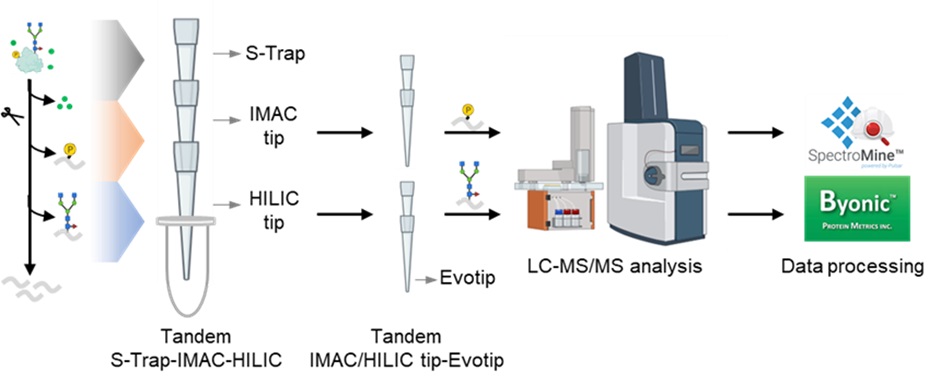
N-糖基化和磷酸化是特別重要的蛋白質轉譯後修飾,在調節植物對環境刺激的反應中發揮著基本作用。然而,當前的同時分析磷酸化蛋白質體和N-糖基化蛋白質體序列富集方法是勞動密集型的,並且耗時,限制了它們的分析通量。為了解決這一挑戰,這項研究引入了一種新穎的串聯固定金屬離子親和層析法與親水交互作用液相層析法策略,稱為TIMAHAC,用於同時分析植物磷酸化蛋白質體學和N-糖基化蛋白質體學。這種方法將固定金屬離子親和層析法和親水交互作用液相層析法整合到串聯微量滴管格式中,通過使用單一的裝載溶液和優化的富集順序來簡化磷酸化胜肽和N-糖基化胜肽的富集流程。TIMAHAC的應用性通過分析阿拉伯芥在離層素處理後的磷酸化蛋白質體和N-糖基化蛋白質體得到了證實。從阿拉伯芥中鑑定出多達1,954個N-糖基化胜肽和11,255個磷酸化胜肽,表明此方法適用於植物組織。我們的結果揭示了TIMAHAC提供了一種全面性的方法來研究植物生物學中複雜的調節機制和蛋白質轉譯後修飾間的相互作用,為深入研究植物信號網絡鋪平了道路。
- Sang, T.; Chen, C.-W.; Lin, Z.; Ma, Y.; Du, Y.; Lin, P.-Y.; Hadisurya, M.; Zhu, J.-K.; Lang, Z.; Tao, W. A; Hsu, C.-C.*; Wang, P.* DIA-based phosphoproteomics identifies early phosphorylation events in response to EGTA and mannitol in Arabidopsis. Mol. Cell. Proteomics 2024, 23, 100804.
- Chen, C.-W.; Lin, P.-Y.; Lai, Y.-M.; Lin, M.-H.; Lin, S.-Y.; Hsu, C.-C.* TIMAHAC: streamlined tandem IMAC-HILIC workflow for simultaneous and high-throughput plant phosphoproteomics and N-glycoproteomics. Mol. Cell. Proteomics 2024, 23, 100762.
- Lin, Y.-H.#; Xu, M.-Y.#; Hsu, C.-C. #; Lee, H.-C.; Damei, F. A.; Lee, H.-C.; Tsai, W.-L.; Hoang, C. V.; Chiang, Y.-R.; Ma, L.-S. Ustilago maydis PR-1-like protein has evolved two distinct domains for dual virulence activities. Nat. Commun. 2023, 14, 5755. #equal contribution.
- Chen, C.-W.; Tsai, C.-F.; Lin, M.-H.; Lin, S.-Y.; Hsu, C.-C.* Suspension trapping-based sample preparation workflow for sensitive plant phosphoproteomics. Anal. Chem., 2023, 95, 12232-12239.
- Kim, T.-W.#; Park, C. H.#; Hsu, C.-C.#; Kim, Y.-W.; Ko, Y.-W.; Zhang, Z.; Zhu, J.-Y.; Hsiao, Y.; Branon, T.; Kaasik, K.; Saldivar, E.; Li, K.; Pasha, A.; Provart, N. J.; Burlingame, A. L.; Xu, S.-L.; Ting, A. Y.; Wang, Z.-Y. Mapping the signaling network of BIN2 kinase using TurboID-mediated biotin labeling and phosphoproteomics. Plant Cell 2023, 35, 975-993. #equal contribution.

Lab Members
陳瑾玟 Chin-Wen Chen
助理專案研發學者 Assistant R&D Scientist (2008.09-)
林佩怡 Pei-Yi Lin
副專案研發學者 Associated R&D Scientist (2022.11-)
陳亭安 Ting-An Chen
研究助理 Research Assistant (2024.02-)
陳柏蒼 Po-Tsang Chen
研究助理 Research Assistant (2025.09-) (農生)
Former Members
蕭証元 Jeng-Yuan Shiau
研究助理 Research Assistant (農生)
連雅苹 Ya-Ping Lien
研究助理 Research Assistant
謝豐如 Feng-Ju Hsieh
研究助理 Research Assistant (農生)
葉家豪 Chia-Hao Yeh
研究助理 Research Assistant (農生)
嚴孝榕 Hsiao-Jung Yen
研究助理Research Assistant (2014.10-2020.12) (農生)
賴映糸 Ying-Mi Lai
研究助理Research Assistant (2021.03-2023.05) (農生)
鄧怡君 Yi-Chun Teng
研究助理Research Assistant (2023.11-2024.02) (農生)
國內
- 2024 優秀質譜技術從業人員 - 台灣質譜學會