謝侑穎 ( Hsieh, Phoebe Yu-Ying )
助研究員
- Molecular and Cellular Biology, Harvard University, USA
- Polymicrobial interaction, Microbial ecology and evolution, Genetics and functional genomics, Experimental evolution
- yhsieh@fredhutch.org
- yhsieh@as.edu.tw
- +886-2-2787-1113 (Office: A330/R407 (temporary Office))
- Lab Website
- ORCID
- Google Scholar

微生物──真菌、細菌與噬菌體──很少單獨生存。
它們持續彼此互動,在不同的生態棲位中合作或競爭,例如土壤、植物體內或動物宿主體內。當多種微生物共同定殖於同一宿主時,微生物間的競爭尤其激烈,因為它們必須面對營養受限及宿主免疫反應的壓力。
這些微生物戰爭如何展開,以及它們如何推動微生物的演化,目前仍所知甚少。在PIE實驗室,我們以會感染植物或人類的真菌與細菌為模型,研究多微生物之間的互動。我們致力於理解這些競爭的分子機制與生物學後果,並探討其如何影響宿主與微生物之間的互動關係。
我們採取自下而上的研究策略──包括功能基因體學與實驗演化──來研究細菌病原體(如感染人類的綠膿桿菌 Pseudomonas aeruginosa,與感染植物的 Pseudomonas syringae)如何與真菌與噬菌體競爭。我們使用體外與體內模型系統,試圖揭示微生物競爭與適應生物壓力的基本原則。我們的長期目標是應用這些知識,工程化微生物群落,或發展用於醫療與農業的有效抗微生物策略。
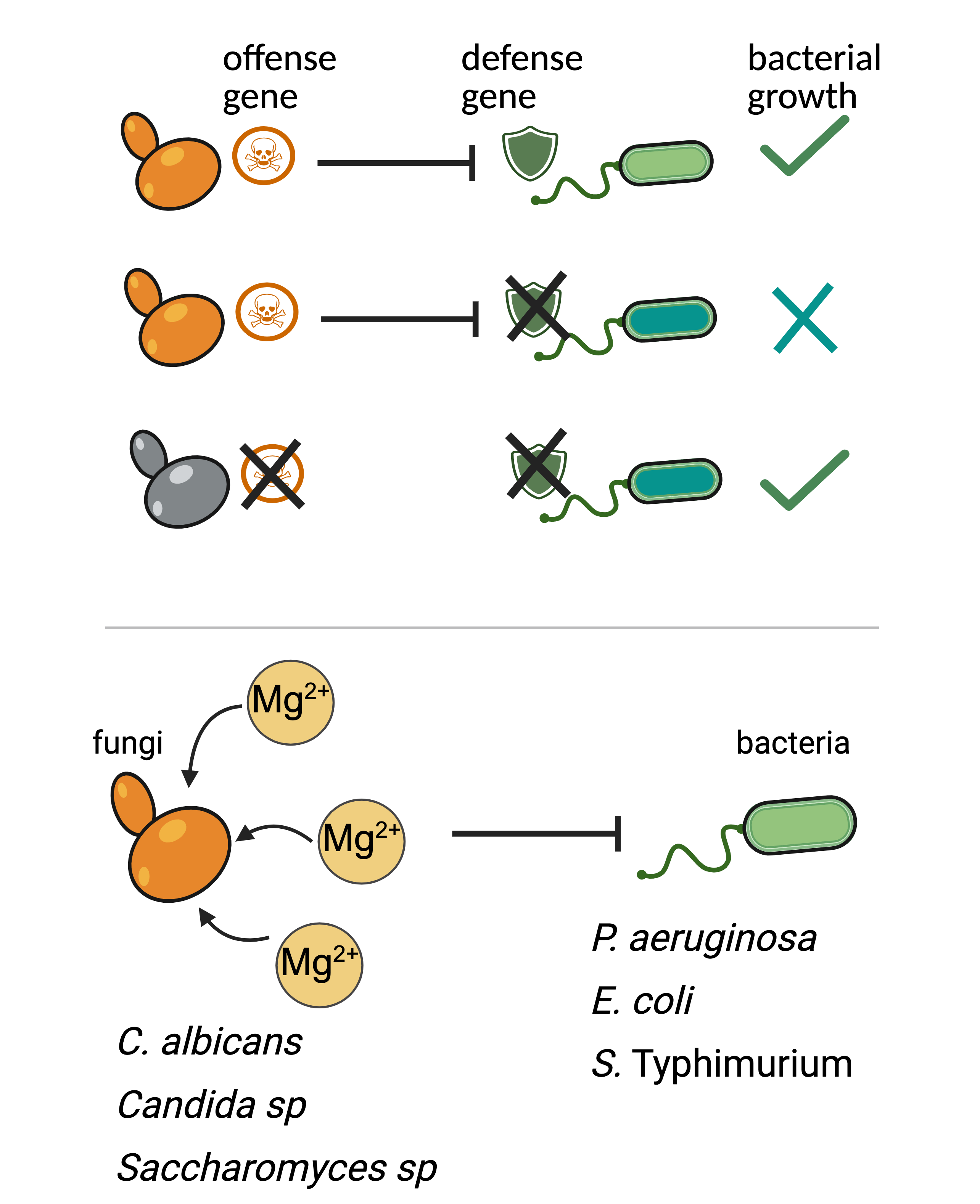
遺傳衝突:真菌與細菌之間的競爭
在多微生物群落中,競爭無可避免。微生物共享相似的生態位,必須演化出策略來爭取資源或空間。雖然細菌間的競爭已有許多研究──通常涉及毒素分泌或營養壟斷──但我們對於細菌如何與真菌競爭仍知之甚少。細菌是否使用不同的分子武器來對抗真菌競爭者?我們又該如何系統性地找出這些機制?
我們將微生物競爭視為一種遺傳衝突:一個物種表達「攻擊」基因來抑制另一物種,而後者則透過閃避或表達「防禦」基因來保護自己。基於這個架構與細菌全基因體適應性篩選的技術,我們先前發現,人類真菌病原 Candida albicans 能藉由螯合鎂離子 (Mg²⁺),抑制人類細菌病原 Pseudomonas aeruginosa 的生長。這種對鎂離子的營養競爭,是多種真菌與革蘭氏陰性菌之間常見的競爭模式。
基於這個發現,我們持續探索其他 Pseudomonas 屬細菌參與真菌競爭的機制,不同真菌如何發展防禦策略,以及在這場軍備競賽中,細菌與真菌如何互相適應演化。

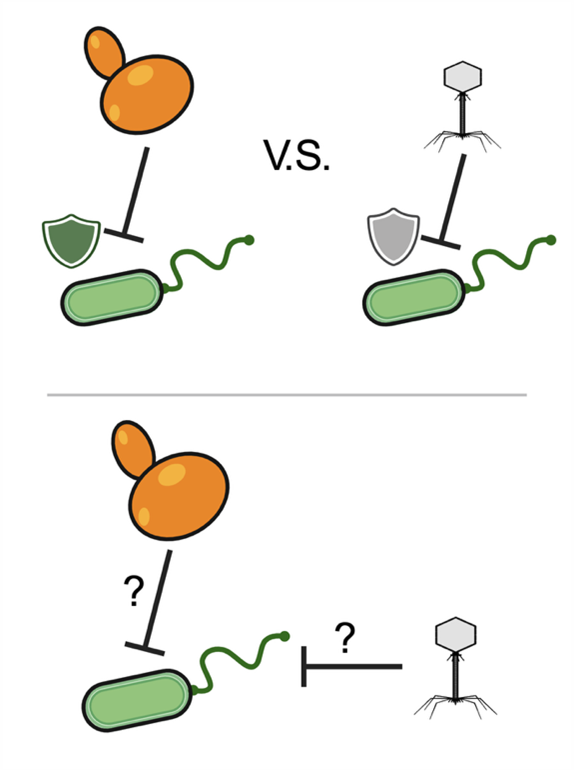
遺傳衝突:真菌-細菌-噬菌體的互動
在多微生物群落中,細菌除了必須與鄰近的真菌競爭外,還得抵抗病毒掠食者──噬菌體。噬菌體會劫持細菌的細胞機器進行複製;而細菌則演化出各種防禦機制來阻止噬菌體進入或複製。與此同時,細菌也可能同時與真菌爭奪資源,甚至進行拮抗。
這引發了關鍵問題:細菌如何同時應對來自真菌與噬菌體的選汰壓力?與真菌的競爭是否會改變細菌對噬菌體的敏感性?反之亦然?我們有興趣理解細菌用來對抗真菌、噬菌體,或同時對抗兩者的防禦基因,以及這些跨界生物衝突如何共同驅動細菌的演化。
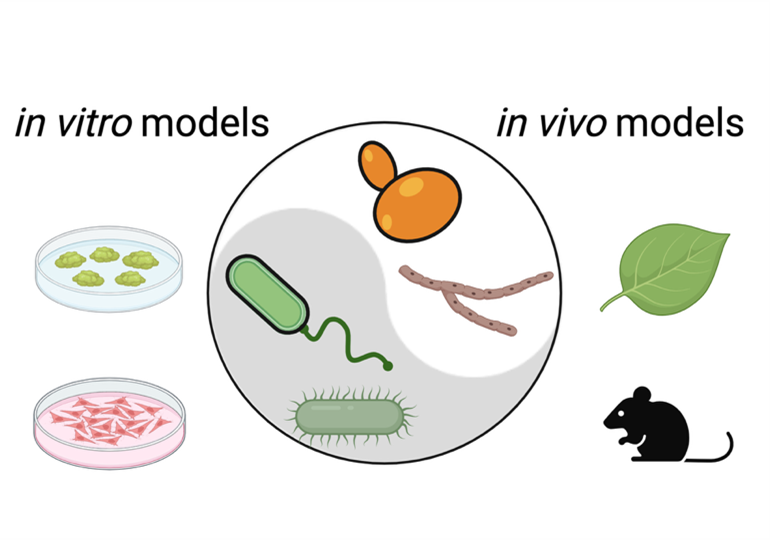
多微生物互動:宿主-微生物交界處的對話
我們先前的研究顯示,真菌與細菌對鎂離子的競爭會影響細菌對抗生素的適應能力,指出這種競爭可能會廣泛影響微生物如何回應其他壓力,例如感染過程中宿主的免疫防禦。
為進一步探討這些影響,我們結合植物與動物感染模型、微生物基因學、基因體學與細胞生物學實驗,找出在多微生物感染中關鍵的真菌與細菌基因,並理解跨界微生物互動如何推動感染疾病的進程與宿主的反應。我們最終的目標,是理解這些互動如何在複雜的宿主相關生態系中,塑造微生物的致病力與適應能力。
Interested in joining us? Contact Phoebe to discuss further project ideas
All publications
Selected Publications
For a full list of Phoebe's publications, click here
- Hsieh, Y. -Y.+*,O’Keefe+, I., Sun, W., Wang, Z. C., Vu, L., Ernst, R., Dandekar, A. A., Malik, H. S. (2024) “Magnesium depletion unleashes two unusual modes of colistin resistance with different fitness costs” BioRxiv preprint DOI: 10.1101/2024.10.15.618514
- Hsieh, Y. -Y.*, Sun, W., Young, J. M., Cheung R., Hogan, D. A., Dandekar, A. A., Malik, H. S.* (2024) “Widespread fungal-bacterial competition for magnesium lowers bacterial susceptibility to polymyxin antibiotics” PLOS BiologyDOI: 10.1371/journal.pbio.3002694
- Hsieh, Y. -Y.*, Makrantoni, V., Robertson D., Marston, A. L., Murray, A. W.* (2020) “Evolutionary repair: changes in multiple functional modules allow meiotic cohesin to support mitosis”
- LaBar T., Hsieh, Y. -Y., Fumasoni M., Murray, A. W.* (2020) “Evolutionary repair experiments as a window to the molecular diversity” Current Biology30: R565-R574 DOI: 10.1016/j.cub.2020.03.046
- Hsieh, Y.-Y., Hung, P.-H., Leu, J-Y.* (2013) “Hsp90 regulates non-genetic variation in response to environmental stress” Molecular Cell50: 82–92 DOI: 10.1016/j.molcel.2013.01.026
- McDonald, M.J., Hsieh, Y.-Y., Yu, Y.-H., Chang, S.-L. and Leu, J.-Y.* (2012) “The evolution of low mutation rates in experimental mutator populations of Saccharomyces cerevisiae” Current Biology22: 1235–1240 DOI: 10.1016/j.cub.2012.04.056