[陳柏仰] 計算全基因組甲基化異質性
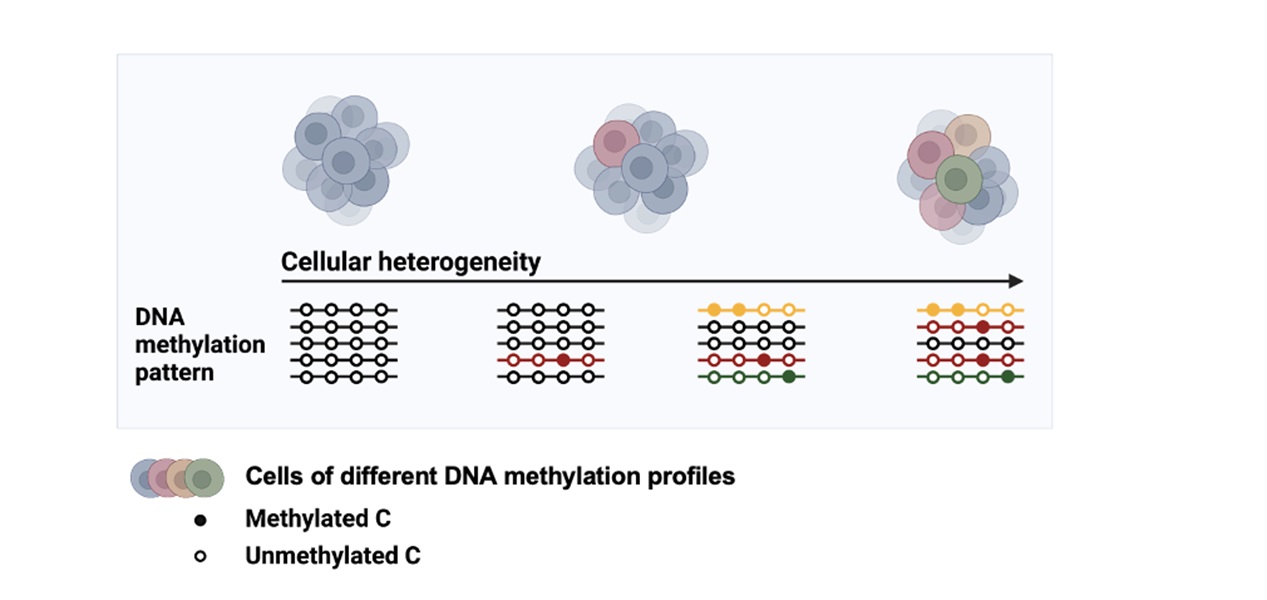
發稿時間:DNA甲基化是生物重要表觀遺傳修飾之一,其中甲基化的異質性(Methylation heterogeneity)可以幫助我們了解細胞發育或是疾病發展的動態變化。甲基化異質性的計算可以透過比較多細胞資料(WGBS、RRBS 和 EM-seq)中每一個細胞的甲基化型態(Methylation pattern)來量化,然而現有的工具大多忽略了甲基化型態間的相似程度以及量化值上升程度的一致性導致明顯誤差。因此 陳柏仰研究室開發了一個計算DNA甲基化異質性的數學模型,此模型改良自生物多樣性模型,將生態系中不同的物種看作是不同的甲基化型態,就可以知道在多細胞資料中甲基化型態的豐富度以及歧異度進而量化甲基化異質性。該數學模型不只考慮了甲基化型態間的相似度,量化值的上升還符合線性的增加,相較於目前所有已發表工具能更精準的描述甲基化的異質性。並且透過分析真實質料,發現了在植物中,CG和non-CG甲基化異質性和甲基化程度之間不同的分佈;在人類大腸癌中,能夠透過找尋甲基化異質性差異位置來找到可以預測疾病發生的生物標記。此數學模型也已經寫成一套軟體MeH,讓使用者可以輸入全基因體甲基化資料,就可以計算出全基因組每個位置的甲基化異質性並找出差異位置與基因。MeH可在 https://github.com/PaoyangLab/MeH 下載使用。