[陳柏仰] MethylC-analyzer: 一個用於分析全基因組 DNA 甲基化的生物資訊工具
發稿時間: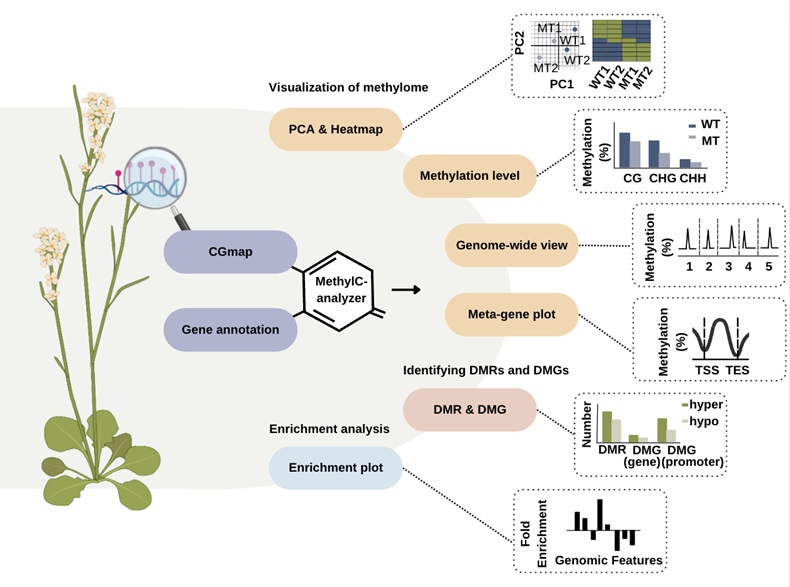
圖: MethylC-analyzer示意圖。 MethylC-analyzer 是用於分析 BS-seq 和 EM-seq 的生物資訊工具。使用MethylC-analyzer需要提供CGmap 和基因註釋文件 (GTF),接下來第一步是生成 CG、CHG 和 CHH的摘要甲基化組圖,包括主成分分析PCA、聚集分析以及每條染色體和以基因為中心的甲基化分佈圖。 也可以進行兩組甲基化基因體間的差異甲基化分析,例如識別甲基化差異位置(DMRs)及其相關基因(DMGs)。 最後會比較特定區域的 DMR 富集狀態並且可視化上述分析。
DNA甲基化是涉及多種生物合成過程和疾病的重要表觀遺傳修飾,其中的非 CG 甲基化(CHG 和 CHH, H 指 A、C 或 T)通常在植物和真菌中調控基因、轉座子並且與發育密切相關。然而相較於現今其他工具多侷限分析CG甲基化程度與差異,陳柏仰研究室開發了MethylC-analyzer用以分析及比較全基因體DNA甲基化,這個生物資訊工具也提供多項視覺化CG 和非CG位置的分析圖表,不限物種且適用WGBS、RRBS 和 EM-seq 的定序資料。MethylC-analyzer 可生成生物樣本的甲基化程度的多種分析圖,尋找甲基化差異位置 (DMR)及相鄰基因並研究甲基化相關的基因組特徵的富集。MethylC-analyzer 是一個基於 Python 和 R 的工具,旨在對甲基化數據進行全面的下游分析,為不熟悉 DNA 甲基化分析的科學家提供一個相對簡易的分析平台。 它既可以作為程式指令,也可以作為 docker 鏡像功能提供給使用者使用,MethylC-analyzer可在http://paoyang.ipmb.sinica.edu.tw/Software.html#SERVICE下載使用。
論文作者共同第一作者陸芮嫻、林蓓郁、嚴明仁為本所研究助理及博士後研究員,吳秉恆為大專實習生。
文章連結:https://as-botanicalstudies.springeropen.com/articles/10.1186/s40529-022-00366-5