[郭志鴻] 以基因體學分析探討植物菌質體16SrI分類群內的物種分界及發展新型分子標記
發稿時間: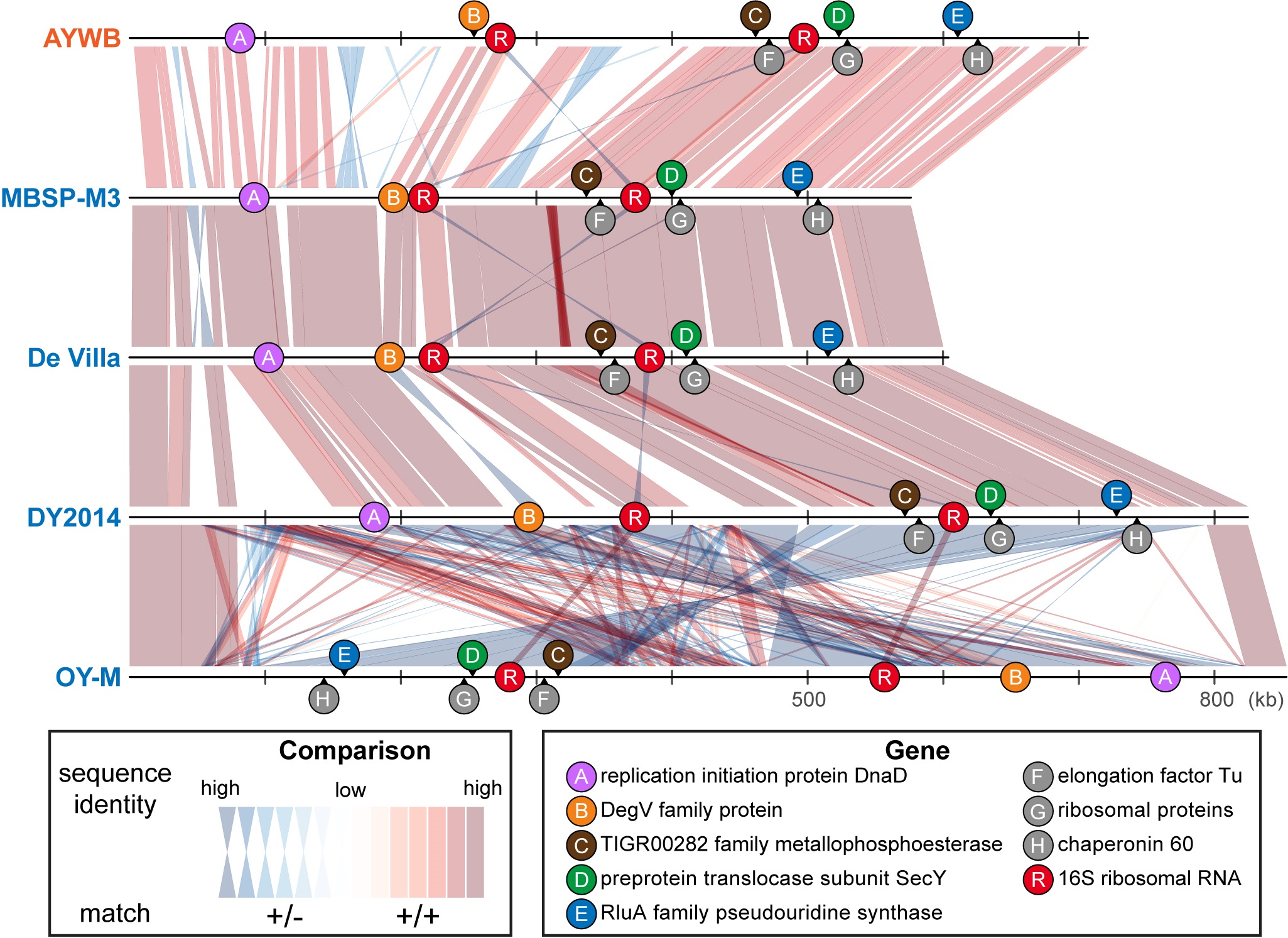
16SrI分類群內不同植物菌質體菌株的全基因體序列排比及分子標記位置。
出處: Cho et al. (2020) Front Microbiol. DOI: 10.3389/fmicb.2020.01531
微生物的分類及鑑定一直是生物學研究中相當基礎且重要的課題。舉例來說,當一個新興的感染性疾病發生時,快速且準確的確認病原是制訂控制策略所需要的關鍵步驟之一。而當感染性疾病發生在不同的地區、族群、宿主,或是引起不同症狀時,確認這些疾病是由相同或是不同的病原所引起也是施行有效控制及進行深入研究時不可或缺的資訊。然而對於難以分離培養的致病性微生物,例如造成農業上重大損失的植物菌質體來說,分類及鑑定數十年來一直面臨種種困難。針對這群至今尚無法被人工培養的植物病菌,目前常用的分類法為1990年代所發展的16S rRNA基因限制片段長度多型性 (restriction fragment length polymorphism; RFLP) 分析法。此方法雖然方便,但卻有一些嚴重的問題,例如單一菌株的基因體內可能帶有兩個被歸在不同分類群的16S rRNA基因,或是不同的菌株帶有極其相似的16S rRNA基因但基因體內其他的基因卻相當不同。也因此這個慣用分類法在準確度跟解析度上都有待改進。
近年來由於DNA定序及基因體學技術的發展,即使是無法被純化培養的細菌也開始可以進行全基因體定序。而因為在農業上的重要性,屬於16SrI分類群的植物菌質體已有超過10個菌株被定序。本計劃利用這組珍貴的資料,針對這群細菌進行全基因體的比較分析。結果發現這些菌株雖然在過去被歸為單一相同物種且所有的16S rRNA基因序列都有超過99% 的相似度,它們基因體內所帶有的基因或是共有基因的序列都有很高的歧異度,也因此可被明確的分為三個不同物種。在同物種內,菌株間共有的基因佔78%以上,且全基因體序列相似度也高達97%以上。而在跨物種的比較上,菌株間共有的基因則在75%以下,全基因體序列相似度也都低於94%。這些發現對基礎的細菌分類學或是具應用性的植物病理學研究都相當重要。
然而在實際應用上,全基因體定序仍然相當費時費力且昂貴,而不可無法被純化培養的細菌則更是難以進行。因此為了要讓研究成果可被拓展到應用層面,本計劃也檢視這些細菌基因體中所有的共有基因,並從中挑選五個鑑別度高的基因開發PCR引子作為分子標記。這五個新型分子標記皆可被單一桑格氏定序法 (Sanger sequencing) 完整解序,且平均散佈於細菌染色體上的不同區域,因此可在維持低成本的前提之下,大幅提升未來植物菌質體田間樣品鑑定的準確度。
此研究由本所郭志鴻博士團隊與英國John Innes Centre之Dr. Saskia Hogenhout團隊合作完成,論文第一作者卓舒婷小姐為郭志鴻老師實驗室的專案經理。此成果發表於國際學術期刊Frontiers in Microbiology,且被收錄於“Mollicutes: From Evolution To Pathogenesis”專題中 (https://www.frontiersin.org/research-topics/9908/mollicutes-from-evolution-to-pathogenesis)。
Cho ST, Kung HJ, Huang W, Hogenhout SA, Kuo CH* (2020) Species boundaries and molecular markers for the classification of 16SrI phytoplasmas inferred by genome analysis. Frontiers in Microbiology 11: 1531. DOI: 10.3389/fmicb.2020.01531