透過機器學習預測阿拉伯芥調控蛋白的協同與拮抗作用
謝誌紘、張雅婷、嚴明仁、謝若微、陳柏仰
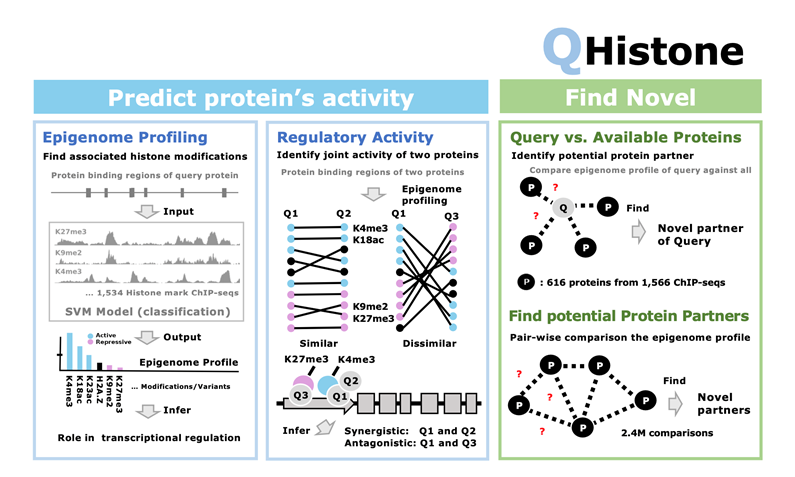
調控蛋白在生物的發育、逆境反應和基因表達調控中扮演著至關重要的角色。而這些蛋白如何通過表觀遺傳機制進行協同或對抗作用,直接影響基因調控的精確性,是表觀遺傳領域中長期待解的核心問題。然而,由於基因調控機制的高度複雜性,實驗技術面臨諸多限制,導致此方向的突破性發現十分稀少。在此研究中,我們通過大量分析約1,500組染色質免疫沉澱測序(ChIP-seq)的全基因體資料,結合機器學習技術,建立了一個高準確性的表觀基因組圖譜,能夠精準預測調控蛋白的功能及其在基因組中的運作規律。此外,我們還比較了約1000個調控蛋白的表觀基因組圖譜,呈現大量前所未知的蛋白質互動關係。值得強調的是,我們的研究在技術與科學發現上具有多項創新。傳統方法難以捕捉的基因間協同或對抗作用,在我們創新的機器學習結合表觀遺傳學的緊密結合下得以精準預測,這填補了實驗技術多年的空白,對了解基因調控網絡提供了前所未有的突破。同時,此成果也為未來以表觀遺傳學為基礎的基因功能研究提供了獨特的視角和工具。為了進一步放大此研究的應用價值,我們開發了 QHistone 網站(https://qhistone.paoyang.ipmb.sinica.edu.tw),為植物科學研究提供了一個公共數據預測分析平台。通過這個平台,即使是不熟悉生物資訊學的研究者,也能輕鬆上傳和解析數據,推動表觀遺傳研究進展。我們的研究不僅在表觀遺傳學理論上有重要突破,也為了植物科學領域帶來了長遠的影響。
Nature Communications, 15(1), 9160.
DOI: 10.1038/s41467-024-53565-y